Lecture des fichiers rates d’Apollo3 : le Reader
La lecture des fichiers rates d’Apollo3 (réseau) est possible dans valjean, ainsi que les différents autres formats HDF5.
Deux modes de lecture sont possibles :
lecture de tout le fichier HDF5 et stockage des résultats dans un
Browser, récupération des résultats sous forme deDatasetgrâce auBrowser-> utilisation duReaderlecture d’un résultat ou de plusieurs résultats donnés à partir du HDF5 pour une utilisation directe sous forme de
Dataset->Picker
La seconde méthode est bien plus rapide car elle ne nécessite pas de charger l’intégralité du fichier et bénéficie des accès de lecture du HDF5.
Cet exemple se concentre sur la première méthode.
Le Reader : résumé
[1]:
from valjean.eponine.apollo3.hdf5_reader import Reader
Lecture d’un fichier rates typique : cas Mosteller, avec isotopes particularisés.
[2]:
ap3r = Reader("full_rates.hdf")
INFO hdf5_reader: Reading full_rates.hdf
INFO hdf5_reader: hdf5 loading done in 0.000725 s
INFO hdf5_reader: Successful reading in 0.013636 s
Transformation en Browser
[3]:
ap3b = ap3r.to_browser()
[4]:
print(ap3b)
Browser object -> Number of content items: 39, data key: 'results', available metadata keys: ['index', 'isotope', 'output', 'result_name', 'zone']
-> Number of globals: 2
39 = nombre de résultats total dans le fichier.
Il est possible de faire les deux précédentes étapes en une seule :
[5]:
from valjean.eponine.apollo3.hdf5_reader import hdf_to_browser
htb = hdf_to_browser("full_rates.hdf")
INFO hdf5_reader: Reading full_rates.hdf
INFO hdf5_reader: hdf5 loading done in 0.000674 s
INFO hdf5_reader: Successful reading in 0.011942 s
Inspection du fichier
On peut inspecter davantage le fichier grâce au Browser et connaître notamment les valeurs possibles des clefs du Browser.
[6]:
print(f'isotopes: {list(ap3b.available_values("isotope"))}')
print(f'resultats: {list(ap3b.available_values("result_name"))}')
print(f'zones: {list(ap3b.available_values("zone"))}')
print(f'outputs: {list(ap3b.available_values("output"))}')
isotopes: ['TotalResidual_reduced_chain', 'I135', 'Sm149', 'U238', 'Xe135', 'macro']
resultats: ['concentration', 'flux', 'absorption', 'diffusion', 'nexcess', 'fission', 'fissionspectrum', 'nufission', 'total', 'current', 'keff', 'kinf', 'production', 'surfflux']
zones: ['q', 'totaloutput']
outputs: ['output_0']
Toutes les combinaisons ne sont pas possibles.
Pour sélectionner un résultat :
si on ne sélectionne qu’un seul résultat : méthode
select_by→ dictionnaire correspondant au résultat demandési on en sélectionne plusieurs en vue d’une sélection plus raffinée ensuite :
filter_by→ sous-Browserdont la liste de résultats a été réduite à ceux correspondant à la sélection demandée
Le résultat, sous forme de Dataset, se trouve sous la clef 'results'.
[7]:
sb_totout = ap3b.filter_by(zone='totaloutput')
print(sb_totout)
print(f'resultats: {list(sb_totout.available_values("result_name"))}')
print(f'outputs: {list(sb_totout.available_values("output"))}')
Browser object -> Number of content items: 7, data key: 'results', available metadata keys: ['index', 'output', 'result_name', 'zone']
-> Number of globals: 2
resultats: ['absorption', 'current', 'flux', 'keff', 'kinf', 'production', 'surfflux']
outputs: ['output_0']
[8]:
keffs = sb_totout.filter_by(result_name="keff")
print(len(keffs))
1
[9]:
keff = sb_totout.select_by(result_name="keff")
print(keff)
{'result_name': 'keff', 'results': class: <class 'valjean.eponine.dataset.Dataset'>, data type: <class 'numpy.float32'>
value: 1.321053e+00, error: nan, bins: OrderedDict()
name: '', what: 'keff'
, 'zone': 'totaloutput', 'output': 'output_0', 'index': 3}
[10]:
sb_q = ap3b.filter_by(zone='q')
print(sb_q)
Browser object -> Number of content items: 32, data key: 'results', available metadata keys: ['index', 'isotope', 'output', 'result_name', 'zone']
-> Number of globals: 2
[11]:
print(f'isotopes: {list(sb_q.available_values("isotope"))}')
print(f'resultats: {list(sb_q.available_values("result_name"))}')
print(f'outputs: {list(sb_q.available_values("output"))}')
isotopes: ['TotalResidual_reduced_chain', 'I135', 'Sm149', 'U238', 'Xe135', 'macro']
resultats: ['concentration', 'flux', 'absorption', 'diffusion', 'nexcess', 'fission', 'fissionspectrum', 'nufission', 'total']
outputs: ['output_0']
[12]:
sb_xe135 = sb_q.filter_by(isotope='Xe135')
print(f'resultats pour Xe135: {list(sb_xe135.available_values("result_name"))}')
resultats pour Xe135: ['concentration', 'absorption', 'diffusion']
[13]:
sb_u238 = sb_q.filter_by(isotope='U238')
print(f'resultats pour U238: {list(sb_u238.available_values("result_name"))}')
resultats pour U238: ['concentration', 'absorption', 'diffusion', 'fission', 'fissionspectrum', 'nexcess', 'nufission']
Sélection des résultats
La liste des résultats est disponible dans content dans le Browser ou directement grâce à la méthode select_by si la sélection est réduite à un résultat unique.
Les résultats sont sotckés sous la clef 'results' sous forme de Dataset.
La plupart des résultats sont donnés par groupes d’énergie. Les intervalles (bins dans le Dataset) correspondent à l’index des groupes.
Certaines quantités ont différents axes :
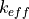 et
et  : grandeurs scalaires, pas d’axes
: grandeurs scalaires, pas d’axesdiffusion : le nombre d’anisotropies sur lequel le calcul est fait est pris en compte, s’il est différent de 1 les axes seront (anisotropies, groupes)
flux surfacique : les axes sont (groupes, surfaces), surfaces contenant l’index des surfaces
courant : les axes sont (groupes, surfaces, direction), surfaces contenant l’index des surfaces et direction (incoming et leaving)
[14]:
xe135_abs = sb_q.select_by(isotope='Xe135', result_name='absorption')
print(list(xe135_abs.keys()))
xe135_abs = xe135_abs['results']
print(xe135_abs.shape) # 26 groupes
print(xe135_abs.what)
xe135_abs.name = '$^{135}$Xe'
['result_name', 'results', 'isotope', 'zone', 'output', 'index']
(26,)
absorption
[15]:
macro_diff = sb_q.select_by(isotope='macro', result_name='diffusion')
print(macro_diff['results'].what)
print(macro_diff['results'].shape)
print(list(macro_diff['results'].bins.keys()))
diffusion
(4, 26)
['anisotropies', 'groups']
[16]:
surf_flux = sb_totout.select_by(result_name='surfflux')
print(surf_flux['results'].what)
print(surf_flux['results'].shape)
print(list(surf_flux['results'].bins.keys()))
surfflux
(26, 220)
['groups', 'surfaces']
[17]:
current = sb_totout.select_by(result_name='current')['results']
print(current.what)
print(current.shape)
print(current.bins)
current
(26, 220, 2)
OrderedDict([('groups', array([ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16,
17, 18, 19, 20, 21, 22, 23, 24, 25])), ('surfaces', array([ 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12,
13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25,
26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38,
39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51,
52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64,
65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 76, 77,
78, 79, 80, 81, 82, 83, 84, 85, 86, 87, 88, 89, 90,
91, 92, 93, 94, 95, 96, 97, 98, 99, 100, 101, 102, 103,
104, 105, 106, 107, 108, 109, 110, 111, 112, 113, 114, 115, 116,
117, 118, 119, 120, 121, 122, 123, 124, 125, 126, 127, 128, 129,
130, 131, 132, 133, 134, 135, 136, 137, 138, 139, 140, 141, 142,
143, 144, 145, 146, 147, 148, 149, 150, 151, 152, 153, 154, 155,
156, 157, 158, 159, 160, 161, 162, 163, 164, 165, 166, 167, 168,
169, 170, 171, 172, 173, 174, 175, 176, 177, 178, 179, 180, 181,
182, 183, 184, 185, 186, 187, 188, 189, 190, 191, 192, 193, 194,
195, 196, 197, 198, 199, 200, 201, 202, 203, 204, 205, 206, 207,
208, 209, 210, 211, 212, 213, 214, 215, 216, 217, 218, 219])), ('direction', array(['incoming', 'leaving'], dtype='<U8'))])
Comparaison de résultats
Les tests disponibles dans valjean peuvent tout à fait être appliqués aux résultats issus du Reader ou du Picker.
Ici TestApproxEqual : les résultats sont des float sans erreur associée (mise à nan par défaut).
Import du test et des représentations (voir les autres notebooks)
[18]:
from valjean.gavroche.test import TestApproxEqual
from valjean.javert.representation import FullRepresenter
from valjean.javert.rst import RstFormatter
from valjean.javert.mpl import MplPlot
from valjean.javert.verbosity import Verbosity
frepr = FullRepresenter()
rstformat = RstFormatter()
Comparaison des aborptions pour différents isotopes
[19]:
sm149_abs = sb_q.select_by(isotope='Sm149', result_name='absorption')['results']
sm149_abs.name = '$^{149}$Sm'
i135_abs = sb_q.select_by(isotope='I135', result_name='absorption')['results']
i135_abs.name = '$^{135}$I'
[20]:
taeq_res = TestApproxEqual(xe135_abs, sm149_abs, i135_abs, name='TestApproxEqual',
description='Test le TestApproxEqual sur les absorptions', rtol=1e-2).evaluate()
print(bool(taeq_res)) # expected: False
False
Ajout d’une fonction pour passer en échelle logarithmique l’axe des ordonnées vu les variations du spectre.
[21]:
from valjean.javert import plot_repr as pltr
def log_post(templates, tres):
pltr.post_treatment(templates, tres)
for templ in templates:
templ.subplots[0].attributes.logy = True
return templates
[22]:
logaeqrepr = FullRepresenter(post=log_post)(taeq_res, verbosity=Verbosity.FULL_DETAILS)
aeqrst = rstformat.template(logaeqrepr[1])
print(aeqrst)
.. role:: hl
.. table::
:widths: auto
====== =========== =========== ========================= =========== ========================
groups $^{135}$Xe $^{149}$Sm approx equal($^{149}$Sm)? $^{135}$I approx equal($^{135}$I)?
====== =========== =========== ========================= =========== ========================
0 331.667 100.186 :hl:`False` 620.565 :hl:`False`
1 635.539 1311.99 :hl:`False` 2837.33 :hl:`False`
2 382.903 2705.19 :hl:`False` 1271.12 :hl:`False`
3 1159.48 15132.2 :hl:`False` 1280.17 :hl:`False`
4 1520.81 25141.5 :hl:`False` 1067.43 :hl:`False`
5 1714.74 27434.2 :hl:`False` 1458.82 :hl:`False`
6 2251.86 28455.9 :hl:`False` 2287.66 :hl:`False`
7 3895.33 47164.8 :hl:`False` 2626.3 :hl:`False`
8 15479.9 181483 :hl:`False` 116.592 :hl:`False`
9 42628.9 490062 :hl:`False` 228.763 :hl:`False`
10 146457 3.32796e+06 :hl:`False` 679.754 :hl:`False`
11 9.26102e+06 7.66008e+06 :hl:`False` 2146.12 :hl:`False`
12 1.89378e+08 492069 :hl:`False` 2541.36 :hl:`False`
13 4.80216e+07 184737 :hl:`False` 245.923 :hl:`False`
14 2.49171e+07 126140 :hl:`False` 111.619 :hl:`False`
15 6.97439e+07 587325 :hl:`False` 271.223 :hl:`False`
16 4.34587e+07 794446 :hl:`False` 146.31 :hl:`False`
17 1.05815e+08 7.34886e+06 :hl:`False` 306.338 :hl:`False`
18 7.63633e+08 1.40516e+07 :hl:`False` 1322.4 :hl:`False`
19 5.2979e+09 4.73993e+06 :hl:`False` 2953.29 :hl:`False`
20 2.02055e+10 1.47097e+07 :hl:`False` 3355.33 :hl:`False`
21 1.57765e+11 1.68924e+08 :hl:`False` 7251.37 :hl:`False`
22 6.50512e+11 1.1291e+09 :hl:`False` 10870 :hl:`False`
23 6.84439e+11 6.02307e+08 :hl:`False` 11398.9 :hl:`False`
24 2.319e+11 1.28561e+08 :hl:`False` 6121.15 :hl:`False`
25 3.42816e+10 1.73609e+07 :hl:`False` 1198.21 :hl:`False`
====== =========== =========== ========================= =========== ========================
[23]:
mpl = MplPlot(logaeqrepr[0]).draw()

Comparaison de la diffusion pour les différentes valeurs d’anisotropie
La méthode squeeze permet de supprimer les dimensions triviales d’un Dataset (on ne comparera que des Datasets à une dimension ici).
[24]:
macro_diff_aniso = [macro_diff['results'][:1, :].squeeze()]
macro_diff_aniso[-1].name = 'anisotropie = 0'
macro_diff_aniso[-1].what = 'diffusion (macro)'
print(macro_diff_aniso[-1])
shape: (26,), dim: 1, type: <class 'numpy.ndarray'>, bins: ['groups: [ 0 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25]'], name: anisotropie = 0, what: diffusion (macro)
[25]:
# anisotropie d'ordre 1
macro_diff_aniso.append(macro_diff['results'][1:2, :].squeeze())
macro_diff_aniso[-1].name = 'anisotropie = 1'
macro_diff_aniso[-1].what = 'diffusion (macro)'
# anisotropie d'ordre 2
macro_diff_aniso.append(macro_diff['results'][2:3, :].squeeze())
macro_diff_aniso[-1].name = 'anisotropie = 2'
macro_diff_aniso[-1].what = 'diffusion (macro)'
# anisotropie d'ordre 3
macro_diff_aniso.append(macro_diff['results'][3:, :].squeeze())
macro_diff_aniso[-1].name = 'anisotropie = 3'
macro_diff_aniso[-1].what = 'diffusion (macro)'
[26]:
taeq_res = TestApproxEqual(*macro_diff_aniso, name='TestApproxEqual',
description=("Test le TestApproxEqual sur la diffusion macroscopique aux différents "
"ordres d'anisotropie"),
rtol=1e-2).evaluate()
print(bool(taeq_res)) # expected: False
False
[27]:
aeqrepr = frepr(taeq_res, verbosity=Verbosity.FULL_DETAILS) # il s'agit d'une liste de templates
aeqrst = rstformat.template(aeqrepr[1])
print(aeqrst)
.. role:: hl
.. table::
:widths: auto
====== =============== =============== ============================== =============== ============================== =============== ==============================
groups anisotropie = 0 anisotropie = 1 approx equal(anisotropie = 1)? anisotropie = 2 approx equal(anisotropie = 2)? anisotropie = 3 approx equal(anisotropie = 3)?
====== =============== =============== ============================== =============== ============================== =============== ==============================
0 3.03941e+13 1.43217e+13 :hl:`False` 8.72864e+12 :hl:`False` 5.80013e+12 :hl:`False`
1 2.12017e+14 9.70531e+13 :hl:`False` 5.52561e+13 :hl:`False` 2.46676e+13 :hl:`False`
2 2.12051e+14 8.9062e+13 :hl:`False` 4.40241e+13 :hl:`False` 1.25229e+13 :hl:`False`
3 6.02824e+14 2.45831e+14 :hl:`False` 1.07446e+14 :hl:`False` 8.16862e+12 :hl:`False`
4 5.65327e+14 2.08306e+14 :hl:`False` 8.53618e+13 :hl:`False` 2.39636e+12 :hl:`False`
5 5.0581e+14 2.12395e+14 :hl:`False` 7.79372e+13 :hl:`False` 1.03219e+11 :hl:`False`
6 4.21794e+14 1.89427e+14 :hl:`False` 7.0032e+13 :hl:`False` 2.68827e+10 :hl:`False`
7 4.03747e+14 1.88263e+14 :hl:`False` 6.97694e+13 :hl:`False` 8.80467e+09 :hl:`False`
8 6.12812e+14 2.84296e+14 :hl:`False` 1.0541e+14 :hl:`False` 6.97243e+09 :hl:`False`
9 5.57152e+14 2.66414e+14 :hl:`False` 9.87652e+13 :hl:`False` 2.07365e+09 :hl:`False`
10 6.82393e+14 3.26437e+14 :hl:`False` 1.21391e+14 :hl:`False` 1.53146e+12 :hl:`False`
11 6.89654e+14 3.37509e+14 :hl:`False` 1.28241e+14 :hl:`False` 7.27767e+12 :hl:`False`
12 3.07237e+14 1.40882e+14 :hl:`False` 5.52886e+13 :hl:`False` 1.30106e+13 :hl:`False`
13 2.30713e+13 1.00686e+13 :hl:`False` 3.84214e+12 :hl:`False` 1.08319e+12 :hl:`False`
14 1.03404e+13 4.49361e+12 :hl:`False` 1.70658e+12 :hl:`False` 4.87285e+11 :hl:`False`
15 2.41663e+13 1.04121e+13 :hl:`False` 3.9247e+12 :hl:`False` 1.13473e+12 :hl:`False`
16 1.25264e+13 5.34194e+12 :hl:`False` 2.00096e+12 :hl:`False` 5.86095e+11 :hl:`False`
17 2.53325e+13 1.07076e+13 :hl:`False` 3.97288e+12 :hl:`False` 1.17618e+12 :hl:`False`
18 9.91162e+13 4.06126e+13 :hl:`False` 1.4576e+13 :hl:`False` 4.41702e+12 :hl:`False`
19 1.87319e+14 7.15993e+13 :hl:`False` 2.33575e+13 :hl:`False` 7.47564e+12 :hl:`False`
20 1.88516e+14 6.65679e+13 :hl:`False` 1.92102e+13 :hl:`False` 7.18871e+12 :hl:`False`
21 3.54498e+14 1.13313e+14 :hl:`False` 2.8835e+13 :hl:`False` 1.29191e+13 :hl:`False`
22 4.7715e+14 1.33495e+14 :hl:`False` 2.8788e+13 :hl:`False` 1.47012e+13 :hl:`False`
23 4.67324e+14 1.06575e+14 :hl:`False` 1.65194e+13 :hl:`False` 9.26162e+12 :hl:`False`
24 2.54186e+14 3.85002e+13 :hl:`False` 2.0665e+12 :hl:`False` 2.26773e+12 :hl:`False`
25 5.54342e+13 3.54245e+12 :hl:`False` -1.4409e+11 :hl:`False` 3.48981e+11 :hl:`False`
====== =============== =============== ============================== =============== ============================== =============== ==============================
[28]:
mpl = MplPlot(aeqrepr[0]).draw()

Comparaison des courants entrants et sortants
Il s’agit d’une comparaison d’arrays en 2 dimensions, mais cela ne change pas les tests.
Pour une question de lisibilité du graphique on se restreint à 20 surfaces.
[29]:
current_in = current[:, :20, :1].squeeze()
current_out = current[:, :20, 1:].squeeze()
[30]:
caeq_res = TestApproxEqual(
current_in, current_out, name='TestApproxEqual',
description=("Test le TestApproxEqual sur la diffusion macroscopique aux différents "
"ordres d'anisotropie"),
rtol=1e-2).evaluate()
print(bool(caeq_res)) # expected: False
False
[31]:
caeqrepr = frepr(caeq_res, verbosity=Verbosity.FULL_DETAILS) # il s'agit d'une liste de templates
caeqrst = rstformat.template(caeqrepr[1])
# print(caeqrst)
[32]:
mpl = MplPlot(caeqrepr[0]).draw()

[33]:
ratio = current_out / current_in
[34]:
import matplotlib.pyplot as plt
import numpy as np
fig, splts = plt.subplots()
bbins = np.broadcast_arrays(ratio.bins['groups'].reshape([26, 1]),
ratio.bins['surfaces'].reshape([20]))
h2d = splts.hist2d(
bbins[0].flatten(), bbins[1].flatten(),
bins=[np.append(ratio.bins['groups']-0.5, [ratio.bins['groups'][-1]+0.5]),
np.append(ratio.bins['surfaces']-0.5, [ratio.bins['surfaces'][-1]+0.5])],
weights=ratio.value.flatten())
cbar = fig.colorbar(h2d[3], ax=splts)
splts.set_xlabel('groups')
splts.set_ylabel('surfaces')
[34]:
Text(0, 0.5, 'surfaces')
