Lecture des fichiers rates d’Apollo3 grâce au Picker
La lecture des fichiers rates d’Apollo3 (réseau) est possible dans valjean, ainsi que les différents autres formats HDF5.
Deux modes de lecture sont possibles :
lecture de tout le fichier HDF5 et stockage des résultats dans un
Browser, récupération des résultats sous forme deDatasetgrâce auBrowser-> utilisation duReaderlecture d’un résultat ou de plusieurs résultats donnés à partir du HDF5 pour une utilisation directe sous forme de
Dataset->Picker
La seconde méthode est bien plus rapide car elle ne nécessite pas de charger l’intégralité du fichier et bénéficie des accès de lecture du HDF5. Cet exemple se concentre sur cette seconde méthode.
Le Picker : résumé
À utiliser notamment si vous connaissez les métadonnées du résultat qui vous intéresse : 'zone', 'output', 'result_name', etc.
[1]:
from valjean.eponine.apollo3.hdf5_picker import Picker
[2]:
ap3p = Picker("Mosteller.hdf")
INFO hdf5_picker: Reading Mosteller.hdf
INFO hdf5_picker: HDF5 loading done in 0.000678 s
La méthode à utiliser pour récupérer les résultats est pick_standard_value :
[3]:
keff = ap3p.pick_standard_value(output='output_1', zone='totaloutput', result_name='KEFF')
Attention : les strings correspondant aux différents arguments (``’output’``, ``’result_name’``, etc) sont celles stockées dans le fichier, en capitales pour les résultats la plupart du temps.
Les résultats correspondants à des taux microscopiques (par isotopes) sont souvent en minuscules avec la première lettre en majuscules.
Inspection du fichier HDF5
Il est tout de même possible d’inspecter le fichier (mais cela peut prendre un peu de temps).
[4]:
print(ap3p.zones(output='output_0'))
print(ap3p.zones(output='output_2'))
['1', '2', '3', 'totaloutput']
['1', '2', '3', 'totaloutput']
Illustration pour l’output 'output_0'.
[5]:
print(f"isotopes dans la zone 3: {ap3p.isotopes(output='output_0', zone='3')}")
print(f"isotopes dans la zone 1: {ap3p.isotopes(output='output_0', zone='1')}")
isotopes dans la zone 3: ['H2O', 'B10', 'B11', 'macro']
isotopes dans la zone 1: ['U234', 'U235', 'U238', 'Pu239', 'Pu241', 'Pu240', 'Pu242', 'O16', 'macro']
Recherche des résultats macroscopiques (pas d’isotope de mentionné) :
[6]:
print(f"résultats dans la zone 1: {ap3p.results(output='output_0', zone='1')}")
print(f"résultats pour totaloutput: {ap3p.results(output='output_0', zone='totaloutput')}")
résultats dans la zone 1: ['FLUX']
résultats pour totaloutput: ['ABSORPTION', 'FLUX', 'KEFF', 'PRODUCTION']
Recherche des résultats macroscopiques et microscopiques (isotope requis) :
[7]:
print(f'resultats sans isotopes: {ap3p.results(output="output_0", zone="3")}')
print(f'resultats pour B10: {ap3p.results(output="output_0", zone="3", isotope="B10")}')
resultats sans isotopes: ['FLUX']
resultats pour B10: ['Absorption', 'concentration']
Sélection des résultats
Récupération d’un résultat sous forme de Dataset : utilisation de la méthode pick_standard_value pour les fichiers rates standards
Exemple sur les 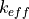
[8]:
keff_o0 = ap3p.pick_standard_value(output='output_0', zone='totaloutput', result_name='KEFF')
print(keff_o0)
value: 1.235028e+00, error: nan, bins: OrderedDict(), type: <class 'numpy.float32'>,name: , what: keff
[9]:
keff_o1 = ap3p.pick_standard_value(output='output_1', zone='totaloutput', result_name='KEFF')
print(keff_o1)
value: 1.247908e+00, error: nan, bins: OrderedDict(), type: <class 'numpy.float32'>,name: , what: keff
[10]:
print(f"Difference between keffs (output_0, output1) = "
f"{(1/keff_o0.value - 1/keff_o1.value)*1e5:.0f} pcm")
Difference between keffs (output_0, output1) = 836 pcm
Exemple sur les flux (arrays)
[11]:
flux = ap3p.pick_standard_value(output='output_0', zone='1', result_name='FLUX')
print(flux)
print(flux.value)
flux.name = 'output 0'
shape: (10,), dim: 1, type: <class 'numpy.ndarray'>, bins: ['groups: [0 1 2 3 4 5 6 7 8 9]'], name: , what: flux
[ 5.168701 45.47962 174.16776 3.8427184 0.37652907
0.4066852 0.73074013 1.4990817 3.1569755 1.5008503 ]
[12]:
absB10 = ap3p.pick_standard_value(output='output_0', zone='3', result_name='Absorption', isotope='B10')
print(absB10)
print(absB10.value)
shape: (10,), dim: 1, type: <class 'numpy.ndarray'>, bins: ['groups: [0 1 2 3 4 5 6 7 8 9]'], name: , what: absorption
[3.4434328e-05 3.5456420e-04 1.6647208e-01 6.4293027e-02 9.4167180e-03
1.5479327e-02 4.2232495e-02 6.3232511e-02 1.6776539e-01 1.6186915e-01]
Comparaison de résultats
Les tests disponibles dans valjean peuvent tout à fait être appliqués aux résultats issus du Reader ou du Picker.
Ici TestApproxEqual : les résultats sont des float sans erreur associée (mise à nan par défaut).
[13]:
flux_1 = ap3p.pick_standard_value(output='output_1', zone='1', result_name='FLUX')
flux_1.name = 'output 1'
[14]:
flux_2 = ap3p.pick_standard_value(output='output_2', zone='1', result_name='FLUX')
flux_2.name = 'output 2'
Import du test et des représentations (voir les autres notebooks)
[15]:
from valjean.gavroche.test import TestApproxEqual
from valjean.javert.representation import FullRepresenter
from valjean.javert.rst import RstFormatter
from valjean.javert.mpl import MplPlot
from valjean.javert.verbosity import Verbosity
frepr = FullRepresenter()
rstformat = RstFormatter()
[16]:
taeq_res = TestApproxEqual(flux, flux_1, flux_2, name='TestApproxEqual',
description='Test le TestApproxEqual sur les flux', rtol=1e-2).evaluate()
print(bool(taeq_res)) # expected: False
False
[17]:
aeqrepr = frepr(taeq_res, verbosity=Verbosity.FULL_DETAILS) # il s'agit d'une liste de templates
aeqrst = rstformat.template(aeqrepr[1])
print(aeqrst)
.. role:: hl
.. table::
:widths: auto
====== =========== =========== ======================= =========== =======================
groups output 0 output 1 approx equal(output 1)? output 2 approx equal(output 2)?
====== =========== =========== ======================= =========== =======================
0 5.1687 5.1609 True 4.78112 :hl:`False`
1 45.4796 45.4164 True 46.6316 :hl:`False`
2 174.168 174.516 True 188.355 :hl:`False`
3 3.84272 3.89218 :hl:`False` 5.57925 :hl:`False`
4 0.376529 0.383804 :hl:`False` 0.87789 :hl:`False`
5 0.406685 0.413455 :hl:`False` 1.69239 :hl:`False`
6 0.73074 0.733685 True 5.25886 :hl:`False`
7 1.49908 1.51683 :hl:`False` 6.91474 :hl:`False`
8 3.15698 3.20994 :hl:`False` 13.5553 :hl:`False`
9 1.50085 1.53149 :hl:`False` 6.58862 :hl:`False`
====== =========== =========== ======================= =========== =======================
[18]:
mpl = MplPlot(aeqrepr[0]).draw()
